近日,中國水產(chǎn)科學研究院黃海水產(chǎn)研究所取得重要科研突破。該所海洋漁業(yè)資源分子生態(tài)學研究團隊成功破譯了鳀魚(學名:日本鳀,Engraulis japonicus)染色體水平基因組,相關(guān)研究成果已發(fā)表于國際期刊《科學數(shù)據(jù)》(Scientific Data)。這項研究為深入理解魚類資源對環(huán)境適應的分子機制,以及科學管理海洋魚類種群提供了重要依據(jù)。

鳀魚 課題組供圖
鳀魚是西北太平洋重要的中上層小型魚類,主要分布在我國渤海、黃海、東海,朝鮮半島沿海以及日本周邊海域。作為黃海、東海食物網(wǎng)的關(guān)鍵物種,鳀魚資源的興衰直接影響藍點馬鮫、帶魚等肉食性經(jīng)濟魚類的餌料供應,在漁業(yè)生態(tài)系統(tǒng)食物鏈中承擔著能量傳遞的"橋梁"作用,具有極高的生態(tài)價值和經(jīng)濟價值。

研究人員展示鳀魚 課題組供圖
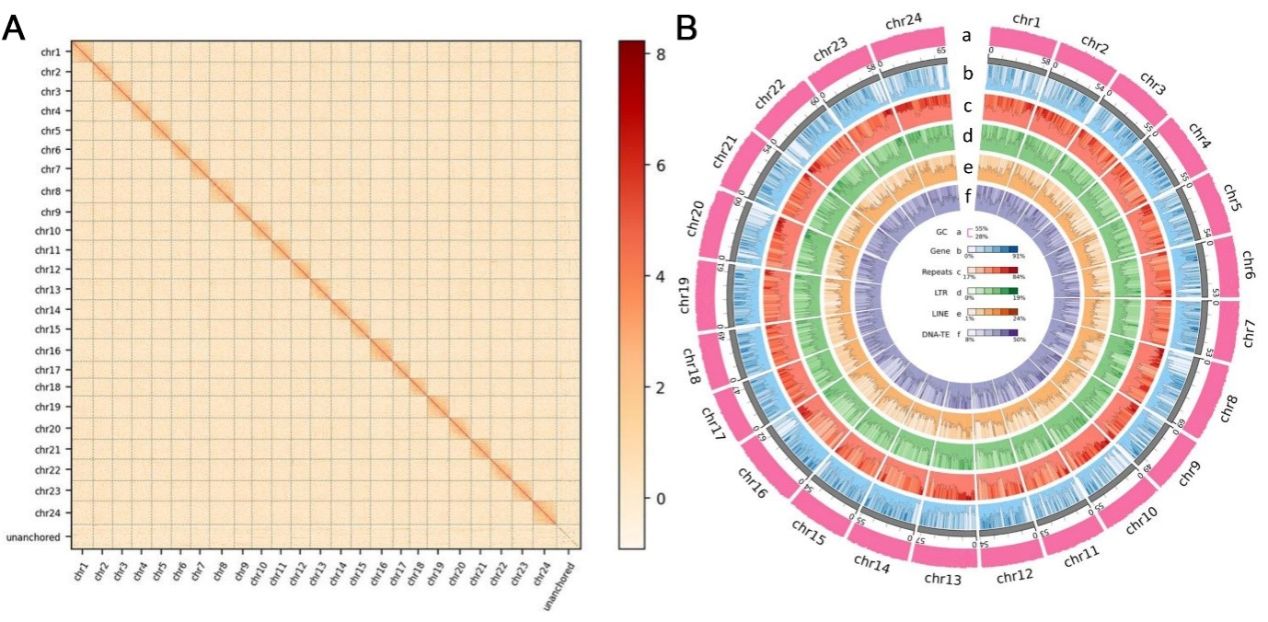
研究團隊采用PacBio高保真長讀長測序技術(shù)、Hi-C染色體構(gòu)象捕獲技術(shù)和Illumina短讀長測序技術(shù),首次構(gòu)建了鳀魚的高質(zhì)量染色體水平基因組。該基因組大小為1467.6兆堿基對,重疊群N50長度為456.3千堿基對,其中1423.3兆堿基對錨定至24條染色體,覆蓋95.2%的組裝序列。
破譯鳀魚染色體水平基因組 課題組供圖
研究結(jié)果顯示,該基因組完整性高達94.07%,共注釋了780.9兆堿基對(54.9%)的重復序列。在重復序列中,長散在核元件(LINE)、短散在核元件(SINE)、長末端重復序列(LTR)和衛(wèi)星DNA序列分別占基因組的6.3%、0.9%、9.1%和2.7%。研究還預測了24,405個蛋白編碼基因,其中97.15%的功能基因獲得數(shù)據(jù)庫注釋,同時預測出23,984個非編碼RNA(ncRNAs)。
黃海水產(chǎn)研究所專家表示,20世紀90年代,我國黃海、東海鳀魚年漁獲量曾高達百萬噸,是當時該海域單一物種資源量最大的魚種。但進入新世紀以來,鳀魚資源急劇衰退,漁獲物呈現(xiàn)低齡化、小型化特征,群體密度持續(xù)下降,目前已難以形成漁汛。鳀魚資源的可持續(xù)利用不僅關(guān)系到區(qū)域漁業(yè)經(jīng)濟發(fā)展,更是維護黃海、東海生態(tài)系統(tǒng)穩(wěn)定的關(guān)鍵所在。
據(jù)悉,這項研究成果為深入挖掘鳀魚遺傳資源、研究小型中上層魚類適應性進化以及開發(fā)資源高值化技術(shù)奠定了重要數(shù)據(jù)基礎(chǔ),將為我國海洋漁業(yè)資源保護和可持續(xù)利用提供科學支撐。
黃海水產(chǎn)研究所研究員柳淑芳和博士王樂為共同第一作者,黃海水產(chǎn)研究所研究員莊志猛為通訊作者。
論文相關(guān)信息:
https://doi.org/10.1038/s41597-025-04423-z
